Download hier de pdf van dit artikel.
De Europese Kaderrichtlijn Water (KRW) verplicht waterbeheerders onderzoek te doen naar de visstand. Om de soortensamenstelling in de rijkswateren in kaart te brengen, maakte Rijkswaterstaat tot voor kort gebruik van (fuiken)vangstgegevens van de commerciële aalvisserij, de passieve vismonitoring. Door het grotendeels verdwijnen van de commerciële aalvisserij op de rijkswateren staat de passieve vismonitoring onder druk. Daarom wordt gezocht naar een alternatieve methode.
eDNA
Een mogelijk alternatief dat Rijkswaterstaat nu onderzoekt, is de eDNA-methode (eDNA = environmental DNA). Deze methode is gebaseerd op het feit dat alle (in het water levende) organismen DNA in hun milieu afscheiden. Dit kan zijn in de vorm van feces, slijm, urine, schubben et cetera. Door het nemen van watermonsters kan dit vrij in het milieu aanwezige DNA ‘gevangen’ worden en op basis van analyse kan vervolgens het aanwezige soortspecifieke DNA aangetoond worden en kan vastgesteld worden van welk organisme het afkomstig is. Met de eDNA-methode wordt alleen de aanwezigheid van vissoorten aangetoond. Over aantallen (per lengteklasse) of biomassa biedt de methode (nog) geen uitsluitsel.
De eDNA-techniek is nog volop in ontwikkeling. Een van de onzekerheden is dat we nog niet precies weten welke inspanning we qua bemonstering moeten leveren om een betrouwbare trefkans te hebben. Een ander punt is dat in het eDNA dat we in stromende wateren aantreffen afkomstig kan zijn van elders. eDNA blijft immers meerdere dagen tot weken intact.
Dit onderzoek is een eerste verkenning naar de toepasbaarheid van de eDNA-methode in grote rivieren. Hiervoor is een vergelijkende veldstudie uitgevoerd, waarbij de resultaten van de reguliere fuikenvisserij vergeleken zijn met die van de eDNA-methode.
Opzet vergelijkende veldstudie
Op een traject in de Gelderse IJssel bij Deventer is de traditionele fuikenvisserij vergeleken met de eDNA-methode. Op drie opeenvolgende dagen is op deze locatie met fuiken gevist en zijn watermonsters genomen. De watermonsters zijn geanalyseerd op DNA van zes vissoorten: twee algemeen voorkomende (baars en blankvoorn), twee minder algemene (winde en alver) en twee zeldzamere (meerval en rivierdonderpad).
In dit onderzoek is gebruik gemaakt van drie stel schietfuiken verspreid over circa 4,5 kilometer. Op hetzelfde traject zijn op vier locaties watermonsters genomen: direct stroomopwaarts van elke fuik en op 1,5 kilometer stroomafwaarts van de meest stroomafwaartse fuik. Om ook de verspreiding van DNA-materiaal over kortere afstanden in kaart te kunnen brengen, zijn op elke locatie in totaal negen watermonsters (van elk 2 liter) genomen, verdeeld over drie raaien op 150 meter afstand van elkaar. Per raai zijn drie watermonsters genomen op plaatsen met een verschillende diepte (één, twee en vijf meter).
De analyses zijn uitgevoerd met de qPCR-methode. Hiermee is het mogelijk om de aanwezigheid en hoeveelheid van (e)DNA aan te tonen. De benodigde gereedschappen – de primers en probes – zijn in dit onderzoek met succes ontwikkeld en gevalideerd.
Resultaten
Van de zes doelsoorten zijn er in de onderzoeksperiode vier in de fuiken aangetroffen (zie tabel 1). Dit is overigens te danken aan het grote aantal fuiken dat werd ingezet. Met één fuik waren we in drie dagen op maximaal drie doelsoorten uitgekomen. Waren alle drie de locaties bemonsterd, maar op slechts één dag dan waren we eveneens op maximaal drie doelsoorten uitgekomen.
Met de eDNA-methode zijn alle zes doelsoorten waargenomen. De aangetroffen eDNA- concentraties verschillen sterk tussen de soorten (zie afbeelding 1). De spreiding van de blankvoorn loopt het meest uiteen, die van de alver het minst.
Tabel 1. De vissoorten (en aantallen) die zijn aangetroffen in de fuiken. De vangsten zijn per dag weergegeven. De codering (F1, F2 en F3) heeft betrekking op de locaties van de fuiken.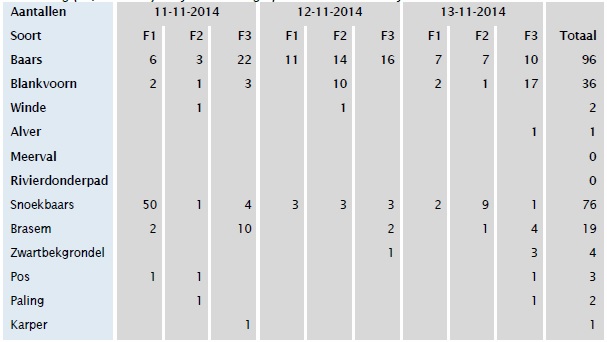
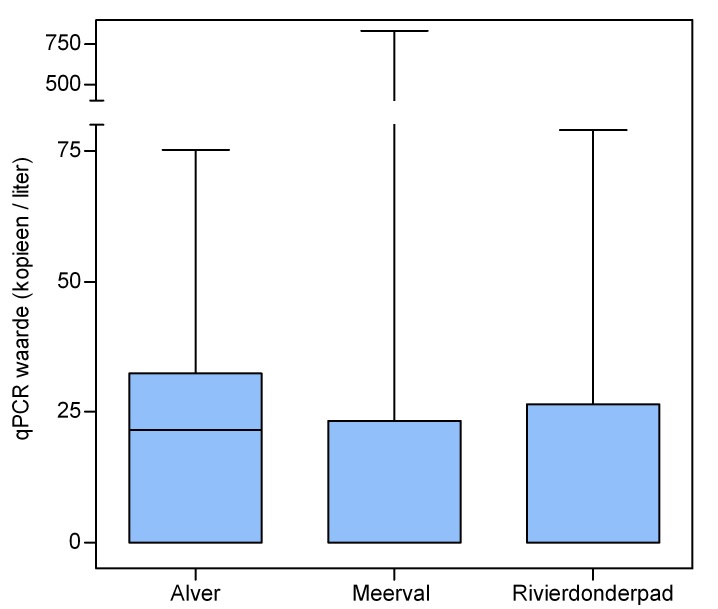
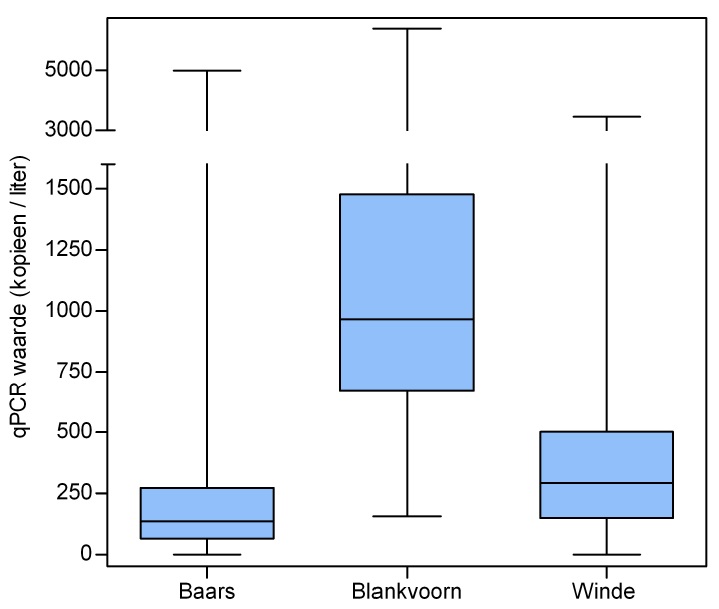 Afbeelding 1. Boxplots van de qPCR-waarden van alle meetpunten over alle dagen per soort. Let op het verschil in schaalverdeling op de y-assen.
Afbeelding 1. Boxplots van de qPCR-waarden van alle meetpunten over alle dagen per soort. Let op het verschil in schaalverdeling op de y-assen.
Wordt in een watermonster eDNA aangetroffen, dan wordt aangenomen dat die soort op het betreffende meetpunt aanwezig is. Zo worden eDNA-verspreidingskaarten gemaakt. Als voorbeeld zijn in figuur 2 en 3 de eDNA-verspreidingskaarten voor baars en meerval weergegeven. Baars is in 97 van de 108 watermonsters aangetoond (90 procent) met qPCR-waarden tot 4.988 kopieën per liter. De verspreiding van meerval-eDNA laat een ander patroon zien. Zo is meerval in slechts 31 van in totaal 108 watermonsters aangetroffen (29 procent) met qPCR-waarden tot 834 kopieën per liter. In de fuikvangsten is geen meerval aangetroffen.
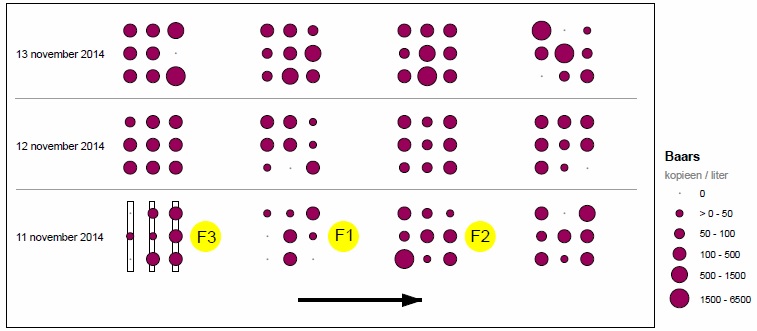
Afbeelding 2. eDNA-verspreidingskaart van baars. Per dag is voor elk meetpunt de gemiddelde waargenomen qPCR-waarde weergegeven. Hoe groter de cirkel, des te meer eDNA in het watermonster is aangetroffen. Per meetlocatie liggen negen meetpunten op drie raaien in een cluster bij elkaar. De meetlocaties liggen ongeveer 1,5 kilometer uit elkaar. De pijl geeft de stromingsrichting aan.
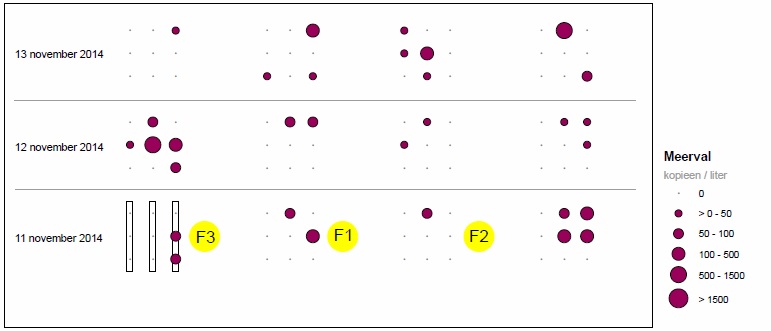 Afbeelding 3. eDNA-verspreidingskaart van meerval. Per dag is voor elk meetpunt de gemiddelde waargenomen qPCR-waarde weergegeven. Hoe groter de cirkel, des te meer eDNA in het watermonster is aangetroffen. Per meetlocatie liggen negen meetpunten op drie raaien in een cluster bij elkaar. De meetlocaties liggen ongeveer 1,5 kilometer uit elkaar. De pijl geeft de stromingsrichting aan.
Afbeelding 3. eDNA-verspreidingskaart van meerval. Per dag is voor elk meetpunt de gemiddelde waargenomen qPCR-waarde weergegeven. Hoe groter de cirkel, des te meer eDNA in het watermonster is aangetroffen. Per meetlocatie liggen negen meetpunten op drie raaien in een cluster bij elkaar. De meetlocaties liggen ongeveer 1,5 kilometer uit elkaar. De pijl geeft de stromingsrichting aan.
De verspreidingskaarten laten zien dat baars en meerval elke dag op elk cluster van negen meetpunten – één meetlocatie – zijn waargenomen. Elke soort die in de fuik is aangetroffen, is ook met eDNA aangetoond. Meerval en rivierdonderpad zijn niet in de fuiken aangetroffen, maar wel met de eDNA-methode.
Uit deze eerste verkenning blijkt dat het mogelijk is om met de eDNA-methode algemeen voorkomende soorten en minder algemeen voorkomende soorten, zoals meerval en rivierdonderpad, aan te tonen. Ten opzichte van de fuikenvisserij geeft de eDNA- methode een grotere kans een vissoort aan te tonen.
Door op meerdere locaties verspreid over het onderzoektraject watermonsters te nemen, hebben we de verspreiding van eDNA – in ruimte en tijd – in kaart kunnen brengen en daarmee zicht gekregen op de trefkans van de doelsoorten. De verspreidingskaarten geven ook inzicht in de herkomst van het eDNA.
Vals negatieve waarnemingen
De verspreiding van eDNA verschilt per soort. Zo is van meerval op veel minder meetpunten eDNA aangetroffen dan van baars. Naarmate eDNA minder verspreid voorkomt in water, neemt de trefkans (de kans dat eDNA van een soort aangetroffen wordt) af. Een soort niet aantonen met eDNA terwijl de soort wél aanwezig is, noemen we een vals negatieve waarneming. Voor meerval is de kans op vals negatieve waarnemingen dus groter dan voor baars.
Inzicht in de soortafhankelijke verspreiding van eDNA in stromende wateren is vereist om een bemonsteringsstrategie te hanteren die de kans op vals negatieve waarnemingen minimaliseert.
Op de eDNA-verspreidingskaarten (zoals afbeeldingen 2 en 3) zien we dat, ongeacht de dag, een willekeurige meetlocatie bemonsterd had kunnen worden om alle zes doelsoorten aan te treffen. De kans op vals negatieve waarnemingen zou bij minder watermonsters per meetlocatie groter zijn geweest, want van de meeste doelsoorten is niet op elk meetpunt eDNA aangetroffen.
Op basis van deze resultaten wordt geconcludeerd dat de trefkans dusdanig hoog is dat de eDNA-methode een geschikt alternatief is voor de passieve vismonitoring.
Vals positieve waarnemingen
Tegelijkertijd moet men er ook op bedacht zijn dat met toenemende trefkansen de kans ook groter wordt dat eDNA van buiten het onderzoektraject wordt opgepikt. Onderzoek toont aan dat eDNA op kilometers afstand van de bron nog gedetecteerd kan worden. De kans op zogeheten vals positieve waarnemingen beïnvloedt de interpretatie van de resultaten en daarmee de toepasbaarheid van de eDNA-methode. Dit geldt bijvoorbeeld voor onderzoek in het kader van de Europese Kaderrichtlijn Water en naar de verspreiding van exoten.
Ook nu bieden de eDNA-verspreidingskaarten uitkomst. Door de hoeveelheid eDNA op twee opeenvolgende meetpunten (of raaien) met elkaar te vergelijken, is de herkomst te herleiden. Voor meerval zien we stroomopwaarts van verschillende meetpunten minder eDNA dan op het meetpunt zelf, wat duidt op een DNA-bron tussen deze twee meetpunten en daarmee de aanwezigheid van meerval.
Conclusies
De belangrijkste conclusies uit dit onderzoek zijn:
- Met de eDNA-methode kunnen we de aanwezigheid van algemene en minder algemene vissoorten op grote rivieren aantonen;
- De eDNA-methode geeft een grotere kans een soort aan te treffen dan de fuikenvisserij;
- eDNA-verspreidingskaarten bieden inzicht in de kans een soort aan te treffen en in de herkomst van eDNA;
- De eDNA-methode is praktisch haalbaar en minder arbeidsintensief dan de fuikenvisserij, en levert toch betrouwbare resultaten;
- De eDNA-methode lijkt een geschikt alternatief voor de passieve monitoring.
Om geschikt te zijn als alternatief voor de passieve monitoring is een betrouwbare, efficiënte en robuuste methode voor alle vissoorten vereist. Hoewel in dit onderzoek algemene en minder algemene soorten zijn opgenomen, is het wenselijk inzicht te krijgen in de trefkans van de gehele visgemeenschap. Hiertoe kan ook de eDNA-metabarcoding-techniek een goede aanvulling vormen. Met behulp van deze niet-kwantitatieve DNA-methode kan een beeld verkregen worden van de totale visgemeenschap. Er blijft echter meer kennis vereist van het gedrag van eDNA in water om zo de kans op vals positieve waarnemingen te verkleinen.
Deze twee onderzoeksvragen kunnen met fundamenteel onderzoek en/of het gebruik van computermodellen beantwoord worden. Dit is echter zeer tijdrovend en arbeidsintensief. Bovendien wordt de hoeveelheid en de spreiding van eDNA in water door veel factoren bepaald, die sterk in ruimte en tijd kunnen variëren. Daarom krijgt een meer pragmatische aanpak de voorkeur met deze verkennende studie als aanzet. Door de combinatie van qPCR en de meetpunten goed te kiezen kan de verspreiding en herkomst van eDNA (onder de heersende milieufactoren) inzichtelijk worden gemaakt. Hierdoor neemt de betrouwbaarheid van de resultaten toe, en wordt fundamenteel onderzoek naar het gedrag van eDNA in stromende wateren minder noodzakelijk.
Dit artikel is ook gepubliceerd in Water Matters van april 2016.
Water Matters is het halfjaarlijkse kenniskatern van H2O.